genose c19: alat skrining "pengendus" covid-19 buatan ugm.
belakangan, alat ini tampak dielu-elukan oleh banyak orang, terutama pemerintah, sebagai terobosan alternatif dalam penanganan pandemi covid. bahkan ada rencana mau diimpor.
is it that good?
[a thread]
belakangan, alat ini tampak dielu-elukan oleh banyak orang, terutama pemerintah, sebagai terobosan alternatif dalam penanganan pandemi covid. bahkan ada rencana mau diimpor.
is it that good?
[a thread]
disclaimer.
komentar dan perspektif dalam thread ini tidak berdasar artikel ilmiah asli yang lengkap, terpublikasi, dan peer-reviewed.
risetnya masih berjalan saat ini.
komentar dan perspektif dalam thread ini tidak berdasar artikel ilmiah asli yang lengkap, terpublikasi, dan peer-reviewed.
risetnya masih berjalan saat ini.
meski demikian, lewat pre-registrasi resmi protokol risetnya (well done for doing this!) dan media massa, cukup banyak informasi yang mulai bisa dipahami dan ditelaah.
tautan: https://clinicaltrials.gov/ct2/show/study/NCT04558372
tautan: https://clinicaltrials.gov/ct2/show/study/NCT04558372
beberapa wawancara/demo dari peneliti:
-
-
-
saya mendapat kesan bahwa para peneliti cukup berhati-hati dalam penyampaiannya dan tidak berusaha mengklaim terlalu dini bahwa risetnya akan berhasil, salut!
-
-
-
saya mendapat kesan bahwa para peneliti cukup berhati-hati dalam penyampaiannya dan tidak berusaha mengklaim terlalu dini bahwa risetnya akan berhasil, salut!
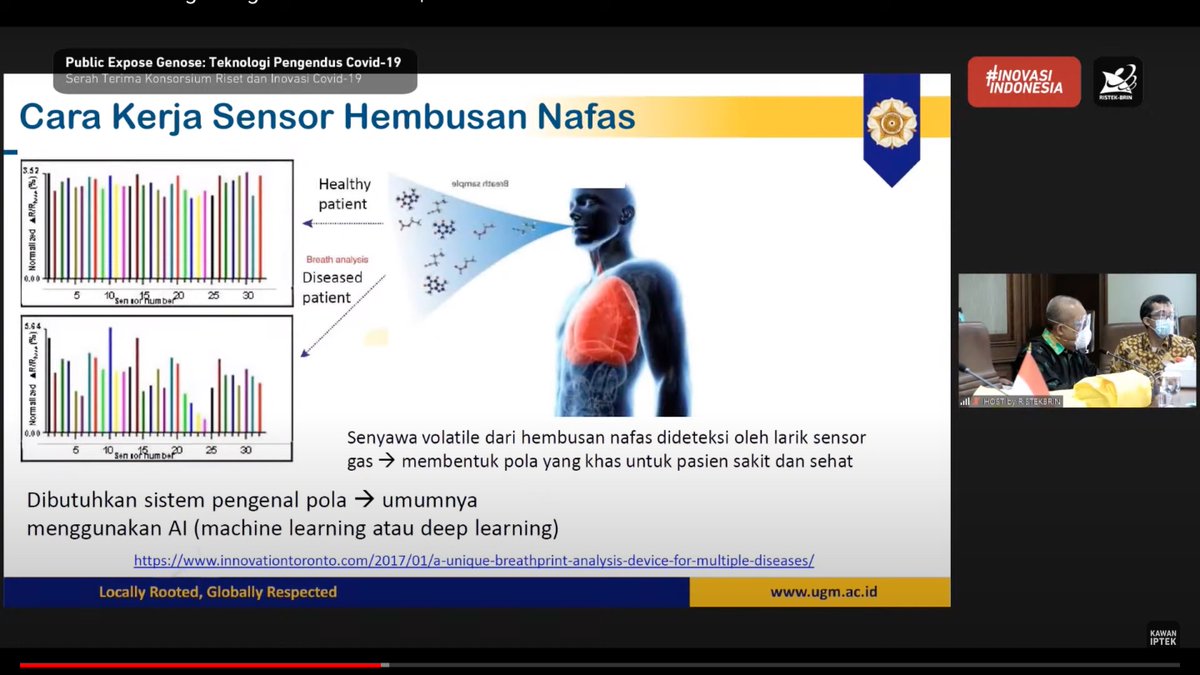
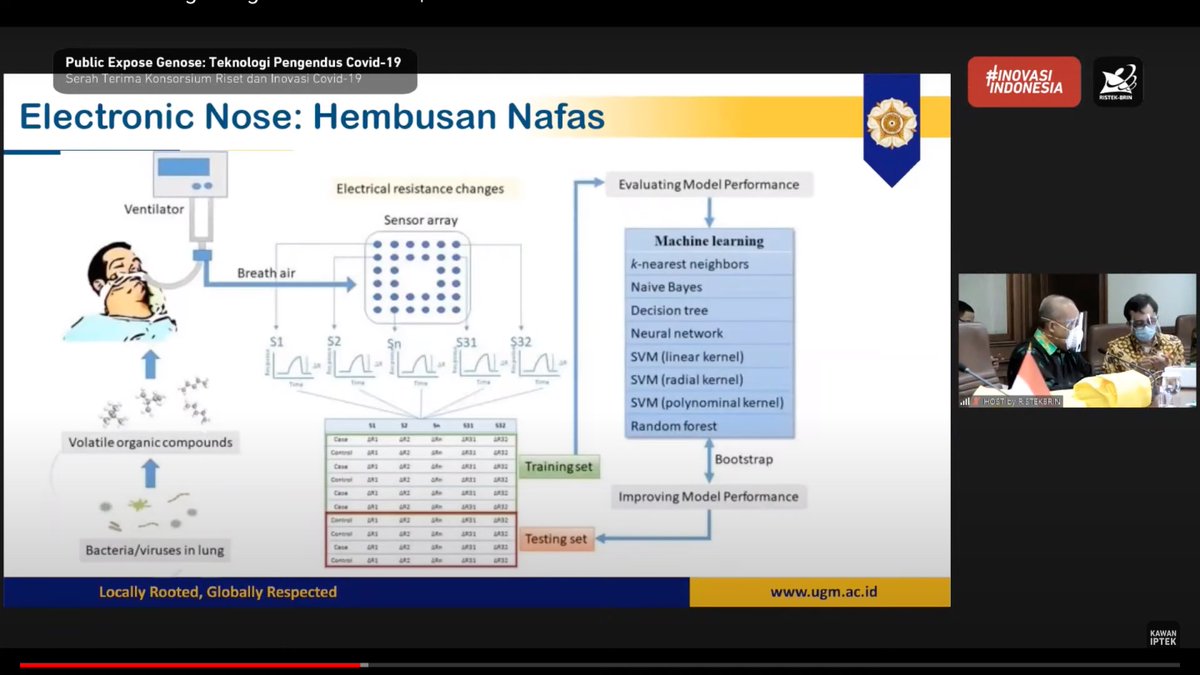
nah, cara kerja alat ini intinya:
1. suspek menghembuskan napas ke air bag
2. sampel napas diteruskan ke alat
3. sensor mendeteksi senyawa-senyawa dalam sampel napas
4. model machine learning (ml) "memahami" *pola senyawa*
5. hasil klasifikasi model: positif/negatif/invalid
1. suspek menghembuskan napas ke air bag
2. sampel napas diteruskan ke alat
3. sensor mendeteksi senyawa-senyawa dalam sampel napas
4. model machine learning (ml) "memahami" *pola senyawa*
5. hasil klasifikasi model: positif/negatif/invalid
jadi, konsepnya berbeda dengan rapid test antigen, antibodi, atau pcr (material genetik virus).
mereka bertiga ini kan berusaha mendeteksi suatu senyawa yang ditarget secara spesifik.
mereka bertiga ini kan berusaha mendeteksi suatu senyawa yang ditarget secara spesifik.
sedangkan genose ini intinya model prediktif, kebetulan berdasar ml.
input modelnya berupa satu atau lebih variabel (dalam hal ini, variabelnya pola-pola senyawa sampel napas).
model ml yang digunakan banyak (lihat kotak biru di gambar), nanti prediksi tiap model digabung.
input modelnya berupa satu atau lebih variabel (dalam hal ini, variabelnya pola-pola senyawa sampel napas).
model ml yang digunakan banyak (lihat kotak biru di gambar), nanti prediksi tiap model digabung.
contoh model prediktif lain yang sudah biasa dipakai di klinis (kebanyakan berdasar outcome regression, bukan ml):
- alvarado score (acute appendicitis)
- apache score (intensive care mortality)
- news2 score (covid-19)
genose ini esensinya sama dengan mereka, suatu model.
- alvarado score (acute appendicitis)
- apache score (intensive care mortality)
- news2 score (covid-19)
genose ini esensinya sama dengan mereka, suatu model.
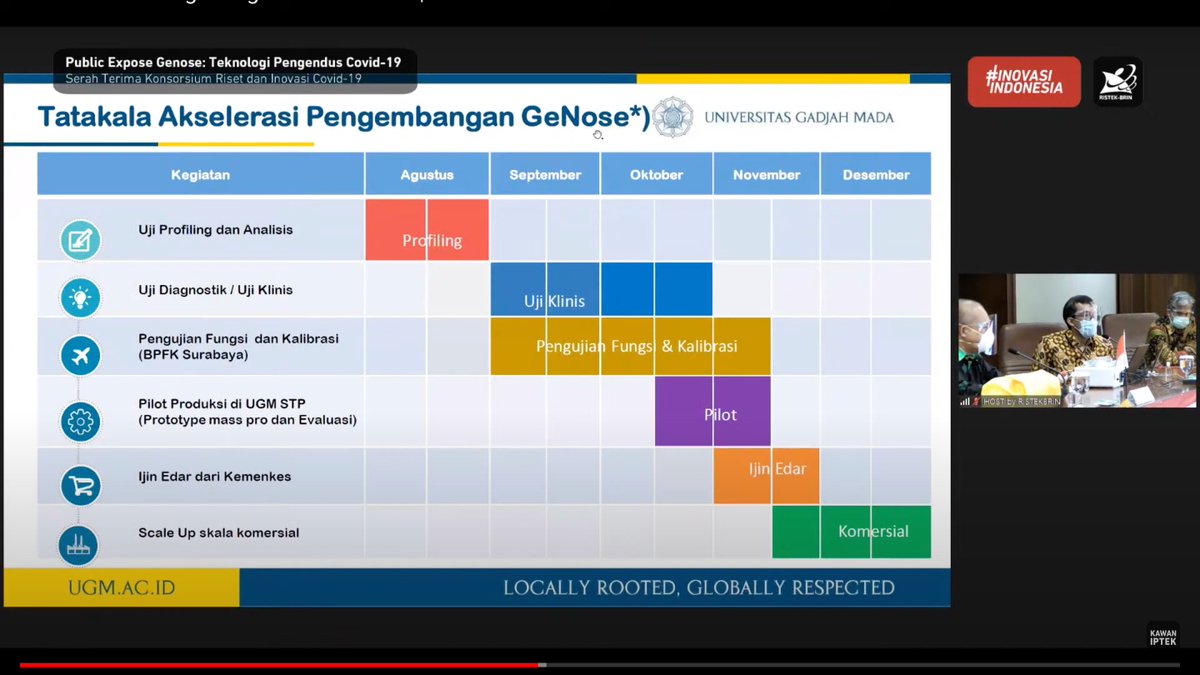
inti riset genose ini ada dua fase:
1. model developement (istilah mereka profiling):
buat machine learning modelnya.
2. external validation (istilah mereka uji diagnostik/klinis):
evaluasi performa akurasi model ke sampel yang representatif.
saat ini sudah masuk fase kedua.
1. model developement (istilah mereka profiling):
buat machine learning modelnya.
2. external validation (istilah mereka uji diagnostik/klinis):
evaluasi performa akurasi model ke sampel yang representatif.
saat ini sudah masuk fase kedua.
oke, semoga cukup buat latar belakangnya.
kalau mau buat critical appraisal lengkap bisa panjang sekali
ada dua pula, model development dan validation. masing-masing punya poin kritik sendiri.
maka dalam thread ini, difokuskan satu isu terpenting:
bias seleksi.
kalau mau buat critical appraisal lengkap bisa panjang sekali

ada dua pula, model development dan validation. masing-masing punya poin kritik sendiri.
maka dalam thread ini, difokuskan satu isu terpenting:
bias seleksi.
pernah dengar hierarchy/pyramid of evidence?
misal nih kita sedang diskusi dalam ranah kedokteran,
bukti ilmiah dari desain penelitian primer apa yang paling baik sebagai dasar evidence-based medicine?
misal nih kita sedang diskusi dalam ranah kedokteran,
bukti ilmiah dari desain penelitian primer apa yang paling baik sebagai dasar evidence-based medicine?
saya cuplik dan highlight dari protokol singkatnya.
termasuk desain studi yang mana riset genose ini?
termasuk desain studi yang mana riset genose ini?
jadi, desain apa yang paling baik? it depends.
tergantung tujuannya apa.
dalam hal uji diagnostik seperti genose ini, desain terbaik untuk menguji performa alat diagnostik adalah...
cross-sectional!
(don't worry if this is unfamiliar to you, keep reading)
tergantung tujuannya apa.
dalam hal uji diagnostik seperti genose ini, desain terbaik untuk menguji performa alat diagnostik adalah...
cross-sectional!
(don't worry if this is unfamiliar to you, keep reading)
kalau dibaca lagi dengan rinci protokolnya, riset genose fase validasi eksternal-nya (fase 2) menurut saya *berpotensi* berkualitas sekali!
biasa kalau sedang kritik paper konklusinya: likely low risk of bias.
biasa kalau sedang kritik paper konklusinya: likely low risk of bias.
contohnya? (yang dalam kurung dampak positifnya)
- multisenter (sampel variatif)
- consecutive sampling (representatif ke superpopulasi)
- triple-blind (less measurement bias)
- 1460 sampel (relatif banyak nih, hasil precise)
jarang hlo begini.
- multisenter (sampel variatif)
- consecutive sampling (representatif ke superpopulasi)
- triple-blind (less measurement bias)
- 1460 sampel (relatif banyak nih, hasil precise)
jarang hlo begini.
sayang, ada tapinya.
tadi dikatakan semua tergantung *tujuannya*. desain riset dan analisis data mengikuti tujuan.
tujuan genose ini buat apa? ternyata..
skrining covid-19 di komunitas, untuk bantu testing-tracing-isolation.
di sini, semua kebaikan di atas, runtuh seketika.
tadi dikatakan semua tergantung *tujuannya*. desain riset dan analisis data mengikuti tujuan.
tujuan genose ini buat apa? ternyata..
skrining covid-19 di komunitas, untuk bantu testing-tracing-isolation.
di sini, semua kebaikan di atas, runtuh seketika.
mengapa?
tidak ada benang merah yang konsisten antara:
- tujuan (skrining covid-19 di komunitas)
- fase 1 (model development)
- fase 2 (external validation)
tidak ada benang merah yang konsisten antara:
- tujuan (skrining covid-19 di komunitas)
- fase 1 (model development)
- fase 2 (external validation)
padahal, inti uji diagnostik itu memastikan representasi pada populasi target (populasi tujuan).
pemilihan subjek penelitian menjadi krusial.
'subject representativeness' di uji diagnostik, imo, kadar pentingnya seperti 'randomisation' di randomised controlled trials!
pemilihan subjek penelitian menjadi krusial.
'subject representativeness' di uji diagnostik, imo, kadar pentingnya seperti 'randomisation' di randomised controlled trials!
siapa populasi targetnya? (ini populasi dimanan alat genose hendak diaplikasikan)
kalau tujuannya untuk skrining dan improve testing-tracing maka minimal:
orang-orang yang kontak erat atau suspek covid-19 di komunitas (general population).
kalau tujuannya untuk skrining dan improve testing-tracing maka minimal:
orang-orang yang kontak erat atau suspek covid-19 di komunitas (general population).
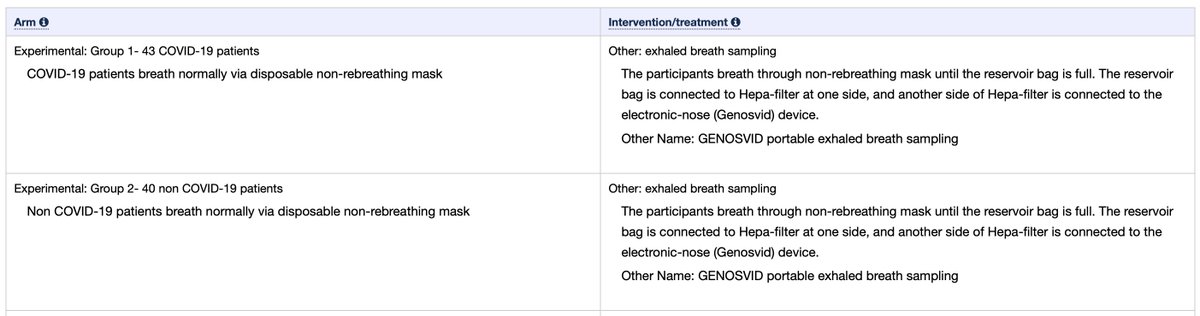
di fase 1 (machine learning model development), populasi apa yang dipakai?
jauh dari populasi target.
hanya sejumlah sampel kecil, diduplikat (non-independen), subjek dari rumah sakit (bukan dari komunitas).
ini juga effectively jadi desain case-control, bukan cross-sectional.
jauh dari populasi target.
hanya sejumlah sampel kecil, diduplikat (non-independen), subjek dari rumah sakit (bukan dari komunitas).
ini juga effectively jadi desain case-control, bukan cross-sectional.
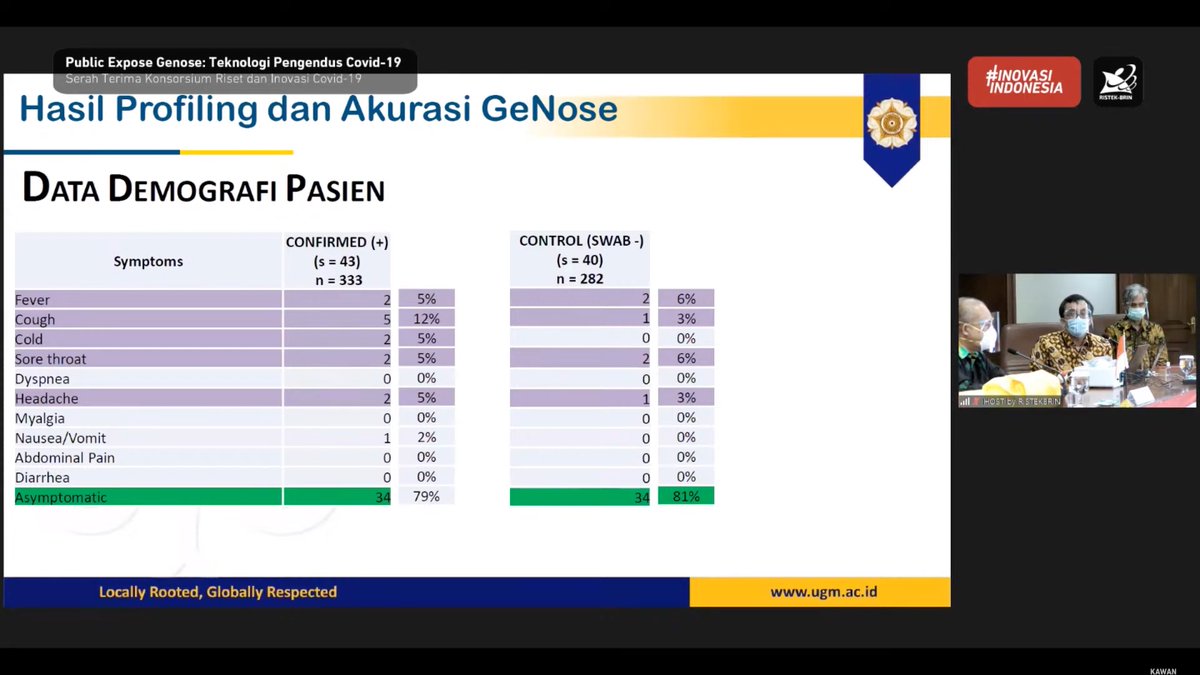
lagi, kalau ada yang familiar dengan predictive model development, jumlah sampel positif (n = 333) dibuat mirip dengan jumlah sampel negatif (n = 282) itu supaya menghindari class imbalance.
biasanya performa model lebih jelek kalau ada class imbalance.
biasanya performa model lebih jelek kalau ada class imbalance.
tapi, di dunia nyata dan praktik klinis..sayangnya, pasti class imbalance!
dalam hal ini, pasien yang dites positif (prevalensi covid-19) pasti rendah.
apalagi kalau ditujukan untuk skrining di komunitas.
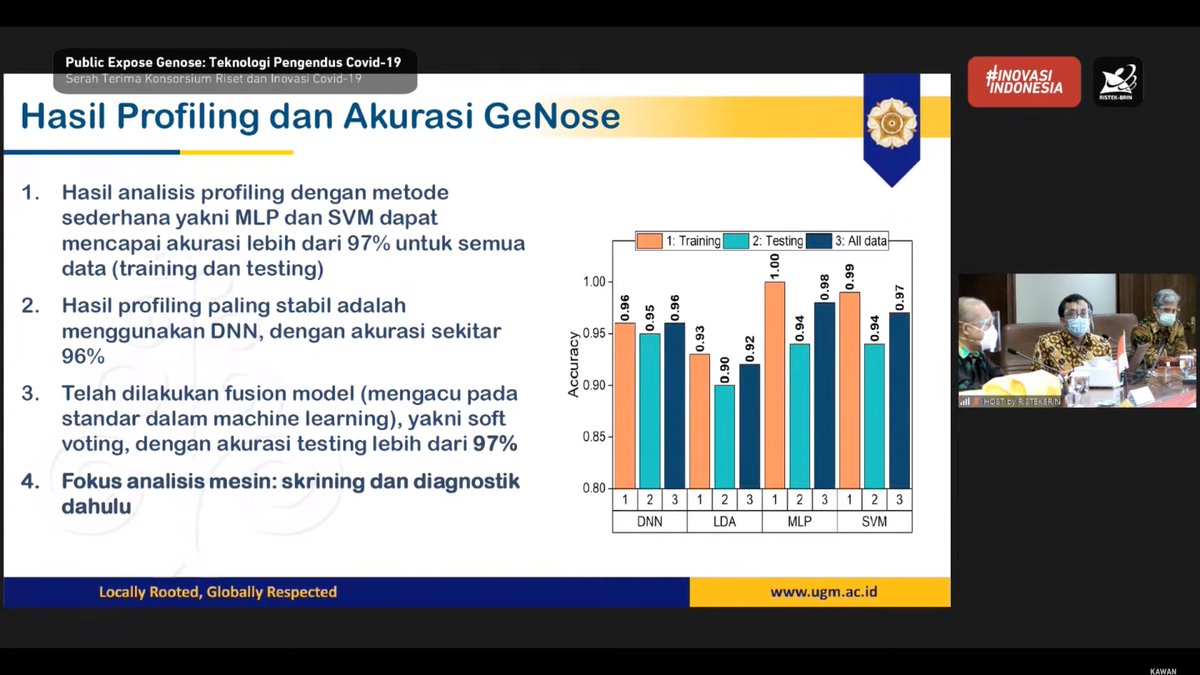
jadi, akurasi 96-97% itu, dipastikan biased performance estimates.
dalam hal ini, pasien yang dites positif (prevalensi covid-19) pasti rendah.
apalagi kalau ditujukan untuk skrining di komunitas.
jadi, akurasi 96-97% itu, dipastikan biased performance estimates.
peneliti ugm sendiri mewanti-wanti kok kalau hasilnya belum final. mereka nyatakan untuk tunggu fase uji diagnostik klinisnya (fase 2).
sayangnya, yang saya pahami dari desain protokol mereka pun, bakal bias juga
bias di fase 2 ini yang sepertinya kurang diapresiasi.
sayangnya, yang saya pahami dari desain protokol mereka pun, bakal bias juga

bias di fase 2 ini yang sepertinya kurang diapresiasi.
populasi apa yang digunakan dalam fase 2?
pasien suspek covid-19 (belum terkonfirmasi) yang datang ke fasilitas rawat jalan rumah sakit.
lebih mendekati memang daripada fase 1, tapi tetap jauh. tidak sesuai dengan populasi tujuan (ie tidak representatif)
pasien suspek covid-19 (belum terkonfirmasi) yang datang ke fasilitas rawat jalan rumah sakit.
lebih mendekati memang daripada fase 1, tapi tetap jauh. tidak sesuai dengan populasi tujuan (ie tidak representatif)
general population dari komunitas itu beda sekali dengan populasi yang datang ke secondary care.
bahkan dengan primary care pun beda (orang yang datang ke puskesmas, praktik pribadi dokter umum).
bahkan dengan primary care pun beda (orang yang datang ke puskesmas, praktik pribadi dokter umum).
di komunitas, prevalensi covid-19 makin rendah, class imbalance makin parah.
predictive value-nya pasti anjlok.
jadi genose ini kondisinya:
model ml-nya sudah tidak dilatih di sampel yang sesuai realitas, divalidasi di sampel yang tidak sesuai tujuan juga.
bias.
predictive value-nya pasti anjlok.
jadi genose ini kondisinya:
model ml-nya sudah tidak dilatih di sampel yang sesuai realitas, divalidasi di sampel yang tidak sesuai tujuan juga.
bias.
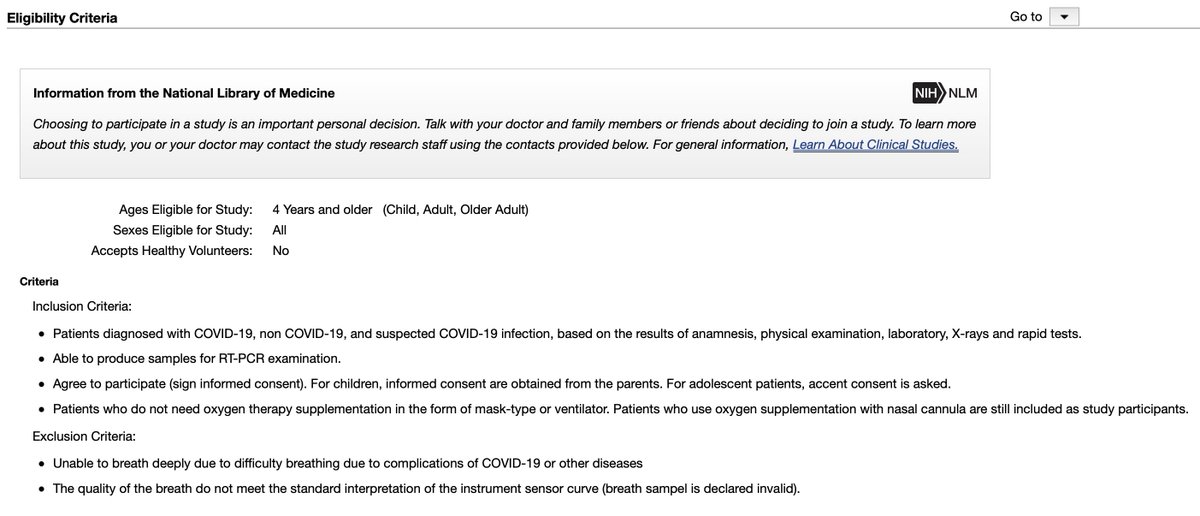
satu lagi terkait sumber bias seleksi lainnya misal: cek yang dihighlight pada kriteria eksklusi.
kalau ada relawan suspek sudah masuk sampel, lalu ternyata kemudian dieksklusi seperti ini, ciri consecutive sampling-nya rusak.
kalau ada relawan suspek sudah masuk sampel, lalu ternyata kemudian dieksklusi seperti ini, ciri consecutive sampling-nya rusak.
bagaimana kalau ternyata kualitas napas yang valid berhubungan dengan prediksi genose dan/atau status covid sesungguhnya? tidak acak.
ini seleksi bias juga kalau hanya menganalisis yang sampelnya valid saja.
jangan dieksklusi, tetap masukkan dalam dataset sebagai missing data.
ini seleksi bias juga kalau hanya menganalisis yang sampelnya valid saja.
jangan dieksklusi, tetap masukkan dalam dataset sebagai missing data.
mengeksklusi subjek yang sudah masuk sampel tuh ibaratnya di randomised controlled trials mirip estimasi analisis per-protocol/as-treated yang likely biased.
padahal mestinya kan intention-to-treat analysis.
ini apaan?haha selingan, lewati saja kalau gak make sense.
padahal mestinya kan intention-to-treat analysis.
ini apaan?haha selingan, lewati saja kalau gak make sense.
demikian contoh mengenai bias seleksi dalam riset genose.
ada beberapa sumber bias lain sebenarnya. tapi bias seleksi yang paling penting.
bisa dibahas kapan-kapan lagi lah kalau ada berita dalam negeri yang menarik dan menyangkut khalayak ramai (monggo saya di-tag saja~).
ada beberapa sumber bias lain sebenarnya. tapi bias seleksi yang paling penting.
bisa dibahas kapan-kapan lagi lah kalau ada berita dalam negeri yang menarik dan menyangkut khalayak ramai (monggo saya di-tag saja~).
oh iya, karena riset genose ini masih ongoing.
beberapa saran yang bisa saya tawarkan untuk meminimalisasi bias seleksi di atas:
beberapa saran yang bisa saya tawarkan untuk meminimalisasi bias seleksi di atas:
1. dengan sampel di fase 2 yang sedemikian rupa, jika memang terbukti performa prediktifnya baik, batasi aplikasinya untuk pasien-pasien suspek covid-19 di rumah sakit saja (paling baik hanya rawat jalan). tidak pada komunitas.
2. hindari dieksklusi relawan yang sudah masuk sampel (terekrut). biarkan dalam dataset sebagai missing data. nanti dilakukan proses imputasi supaya representativeness sampel terhadap populasi rawat jalan terjaga.
3. ini belum sempat dibahas sih. terkait metrik utama performa yang akan dilaporkan.
ini tuh semacam ukuran seberapa "akurat" alatnya.
dengan dasar model ml, justru tidak perlu melaporkan sensitivitas, spesifisitas, positive dan negative predictive values (ppv, npv).
ini tuh semacam ukuran seberapa "akurat" alatnya.
dengan dasar model ml, justru tidak perlu melaporkan sensitivitas, spesifisitas, positive dan negative predictive values (ppv, npv).
keempat metrik di atas perlu dikotomisasi, padahal model ml-nya menghasilkan luaran prediksi berupa probabilitas!
probabilitas tanpa kategorisasi lebih justru meaningful.
analoginya, seperti pcr. yang dilaporkan justru Ct (cycle threshold) nya alih-alih katakan positif/negatif.
probabilitas tanpa kategorisasi lebih justru meaningful.
analoginya, seperti pcr. yang dilaporkan justru Ct (cycle threshold) nya alih-alih katakan positif/negatif.
jadi tidak perlu dikategorisasi jadi positif negatif.
nanti pelaporan performanya pakai apa?
utamanya justru..
- auc: area under the receiver operating curve
- calibration plots
nanti pelaporan performanya pakai apa?
utamanya justru..
- auc: area under the receiver operating curve
- calibration plots
sensitivitas, spesifisitas, ppv, npv boleh sebagai secondary estimates saja di threshold yang dianggap clinically meaningful.
karena tiap klinisi dan pasien sendiri punya threshold value dan pertimbangan risk-benefit yang berbeda.
biarkan dalam bentuk estimated probability.
karena tiap klinisi dan pasien sendiri punya threshold value dan pertimbangan risk-benefit yang berbeda.
biarkan dalam bentuk estimated probability.
4. acknowledgement relawan dalam studi genose sebaiknya ditaruh paling atas sebagai kontributor penting dalam riset ini 

congrats buat peneliti ugm!
if corrected properly, this could be one of the best studies ever done in indonesia during the pandemic!
dan saya sendiri bisa salah dalam interpretasi mengingat keterbatasan ilmu dan informasi yang ada.
i'm open to criticism and further discussion~
if corrected properly, this could be one of the best studies ever done in indonesia during the pandemic!
dan saya sendiri bisa salah dalam interpretasi mengingat keterbatasan ilmu dan informasi yang ada.
i'm open to criticism and further discussion~
untuk uji diagnostik (kecuali sample size calculation) kita tidak perlu mengetahui prevalensi covid di suatu populasi target.
selection bias dalam studi diagnostik dihindari dengan:
1. mensampel subjek yang sesuai dengan populasi target i.e. (cont'd) https://twitter.com/yulianiayi/status/1320180107357483010?s=20
selection bias dalam studi diagnostik dihindari dengan:
1. mensampel subjek yang sesuai dengan populasi target i.e. (cont'd) https://twitter.com/yulianiayi/status/1320180107357483010?s=20
(cont'd) kalau ditargetkan untuk general population, sampelnya harus dari general population.
2. cara samplingnya either 'consecutive' atau 'random dari consecutive'.
riset genose ini terutama tidak sesuai di poin pertama. (cont'd) https://twitter.com/yulianiayi/status/1320180107357483010?s=20
2. cara samplingnya either 'consecutive' atau 'random dari consecutive'.
riset genose ini terutama tidak sesuai di poin pertama. (cont'd) https://twitter.com/yulianiayi/status/1320180107357483010?s=20
(cont'd) dan hampir tepat di poin kedua (karena consecutive sampling), tapi masih ada *potensi* kelemahan karena masih by design mengeksklusi partisipan yang sudah terekrut (jadi tidak purely consecutive lagi karena missing data bias). https://twitter.com/yulianiayi/status/1320180107357483010?s=20
(cont'd) tapi ini selection bias karena missing data belum tentu membuat bias ya.
kalau ternyata cuma satu dua orang, tidak akan bias (not of meaningful magnitude).
atau..kalau ternyata mekanisme missing datanya acak (missing completely at random), tidak akan selection bias.
kalau ternyata cuma satu dua orang, tidak akan bias (not of meaningful magnitude).
atau..kalau ternyata mekanisme missing datanya acak (missing completely at random), tidak akan selection bias.
(cont'd) tapi paling aman, by design, mestinya tidak perlu dieksklusi.
agreed!
btw, saya aware sebelum nulis thread ini kalau para penelitinya punya track record riset uji diagnostik sebelumnya
ini sekadar evaluasi berdasar protokolnya. soalnya tidak bisa analisis tiap observasi jika by design sudah dieksklusi di depan. https://twitter.com/damakusuma/status/1320183109740998656?s=20
btw, saya aware sebelum nulis thread ini kalau para penelitinya punya track record riset uji diagnostik sebelumnya

ini sekadar evaluasi berdasar protokolnya. soalnya tidak bisa analisis tiap observasi jika by design sudah dieksklusi di depan. https://twitter.com/damakusuma/status/1320183109740998656?s=20
mengapa kita tidak seharusnya mengeksklusi sampel yang ‘sulit’ seperti ini?
lagi-lagi representasi. dalam praktik klinis realitasnya, semua keruwetan bisa terjadi dan studi validasi harus bisa menangkap kondisi ini juga (cont’d) https://twitter.com/yulianiayi/status/1320181613817331712
lagi-lagi representasi. dalam praktik klinis realitasnya, semua keruwetan bisa terjadi dan studi validasi harus bisa menangkap kondisi ini juga (cont’d) https://twitter.com/yulianiayi/status/1320181613817331712
(cont’d) analogi, tes pcr.
apa sih yang bikin tes pcr false positive?
- viral load rendah
- salah teknis pas ambil sampel
- medium transport jelek
- salah penanganan pas di lab, dll https://twitter.com/yulianiayi/status/1320181613817331712
apa sih yang bikin tes pcr false positive?
- viral load rendah
- salah teknis pas ambil sampel
- medium transport jelek
- salah penanganan pas di lab, dll https://twitter.com/yulianiayi/status/1320181613817331712
(cont’d) studi validasi klinis pcr yang baik bisa menangkap hal-hal nyebelin tapi memang kenyataan di atas.
maka validasi pcr yg dievaluasi di dalam lab tanpa memikirkan kemungkinan sampel pcr juga bs ‘invalid’ karena poin di atas jadi tidak lagi representatif di praktik klinis. https://twitter.com/yulianiayi/status/1320181613817331712
maka validasi pcr yg dievaluasi di dalam lab tanpa memikirkan kemungkinan sampel pcr juga bs ‘invalid’ karena poin di atas jadi tidak lagi representatif di praktik klinis. https://twitter.com/yulianiayi/status/1320181613817331712
more properly. i should have said ‘false negative’ haha intinya predictive error. https://twitter.com/fadilify/status/1320208846959292416
pembanding modelnya hasil pcr dari tiap individu (sebagai reference/gold standard).
ketika analisis data performa diagnostik, ga perlu tau sih prevalensi aslinya berapa.
justru juga ketika analisis (kalau seleksi relawan baik), otomatis akan tau nanti prevalensinya berapa. https://twitter.com/yulianiayi/status/1320576591567548416
ketika analisis data performa diagnostik, ga perlu tau sih prevalensi aslinya berapa.
justru juga ketika analisis (kalau seleksi relawan baik), otomatis akan tau nanti prevalensinya berapa. https://twitter.com/yulianiayi/status/1320576591567548416

 Read on Twitter
Read on Twitter![genose c19: alat skrining "pengendus" covid-19 buatan ugm.belakangan, alat ini tampak dielu-elukan oleh banyak orang, terutama pemerintah, sebagai terobosan alternatif dalam penanganan pandemi covid. bahkan ada rencana mau diimpor.is it that good?[a thread] genose c19: alat skrining "pengendus" covid-19 buatan ugm.belakangan, alat ini tampak dielu-elukan oleh banyak orang, terutama pemerintah, sebagai terobosan alternatif dalam penanganan pandemi covid. bahkan ada rencana mau diimpor.is it that good?[a thread]](https://pbs.twimg.com/media/ElH9bQeX0AYGu7n.jpg)